分子生物学や生命工学の実験で多用される、制限酵素や電気泳動についてまとめていきましょう。入試問題でも普通に見られるようになりましたね。
ちなみに管理人は、大学院時代は毎日のようにPCRや制限酵素反応、電気泳動をやっていました。当時のことを思い出しながらこの記事を書いています。
PCRについてはこちらもご覧ください。
制限酵素
制限酵素とは、DNAの特定の配列を認識して、その特定の配列でDNAを切断するという特徴をもった酵素です。
よく”ハサミ”の役割を果たすと表現されたりもしますね。
酵素の種類としては、ヌクレオチドを分解する酵素になります。その為、ヌクレアーゼに分類されます。
酵素のうち、
分子を末端から切断・分解していくものをエキソ型、
分子を内側から切断・分解していくものをエンド型と呼びます。
制限酵素はDNAを内側から切断しますので、エンド型のヌクレアーゼということになります。
「制限酵素」という名前の由来が気になる方も多いのではないでしょうか?
研究者たちは、ある種の細菌が外部からのDNAの侵入を”制限”していることを発見しました。
例えばバクテリオファージのように細菌に感染するウイルスは、細菌内に自身のDNAを注入して感染します。それに対する手段として、最近は侵入したファージDNAを分解するシステムを持っており、それが制限酵素であったという訳です。
こうした由来から、ウイルスなどの外部由来のDNAを制限する、という意味を込めて「制限酵素」と名付けられました。
様々な細菌から発見されており、見つかった細菌の名前にちなんで制限酵素も名前がついています。
制限酵素の名前について、EcoRⅠを例に説明してみましょう。
これは大腸菌(Eschericha coli)のRY3という菌株から1番目に発見された制限酵素です。
赤文字を抜き出していくと、EcoRⅠになりますね。このように、「由来となった生物名+発見された順番」というルールで命名されています。
制限酵素の切り方
前述したとおり、特定の配列を認識してその部分を切断するのですが、酵素の種類によって切断する対象となる塩基配列や切断の仕方が異なるという特徴もあります。
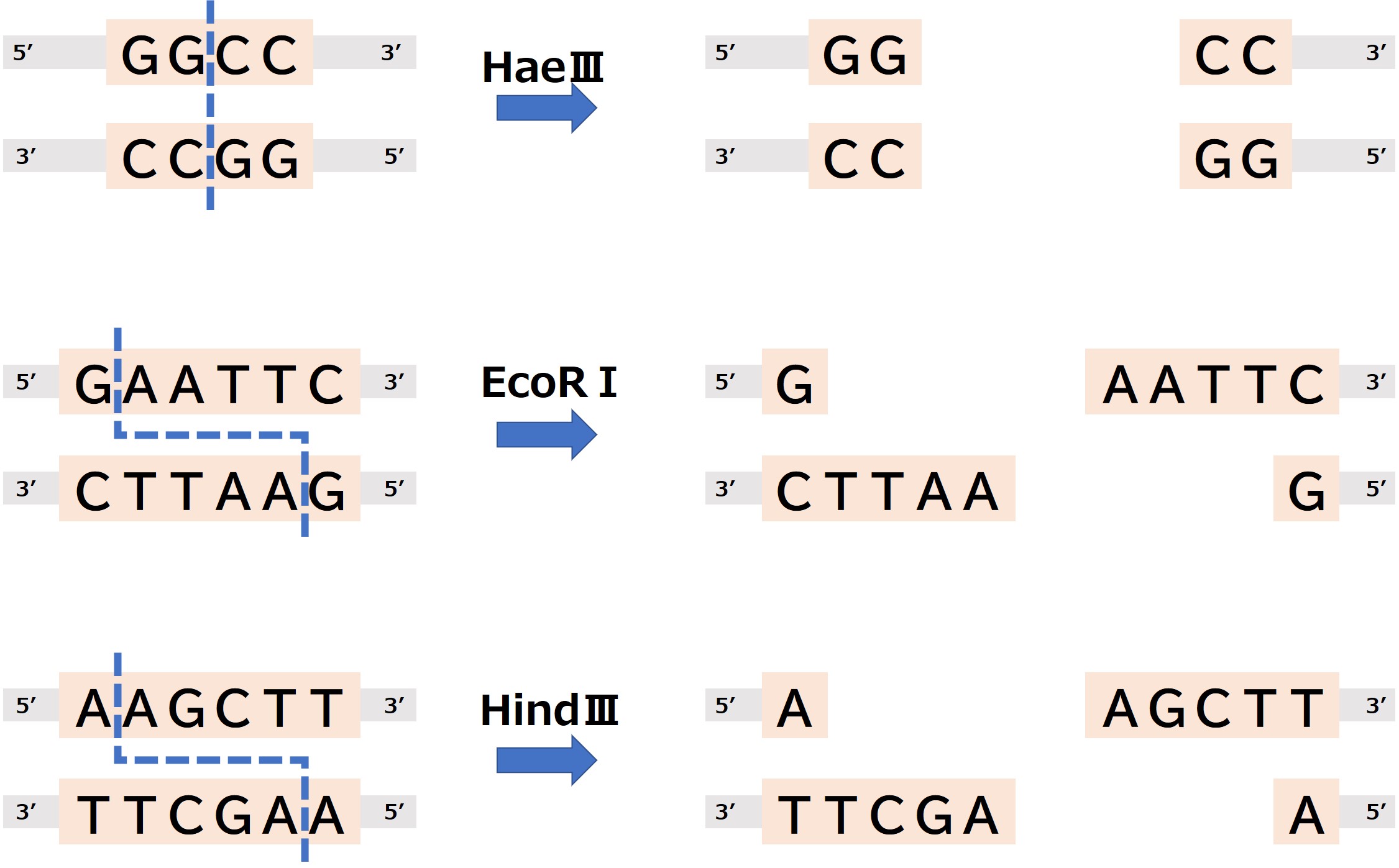
制限酵素が切断する標的とする塩基配列は通常4から8塩基対で、例えばHaeⅢという制限酵素は4塩基対の配列を認識して切断します。
EcoRⅠやHindⅢという制限酵素は、それぞれ異なる6塩基対の配列を認識して切断します。
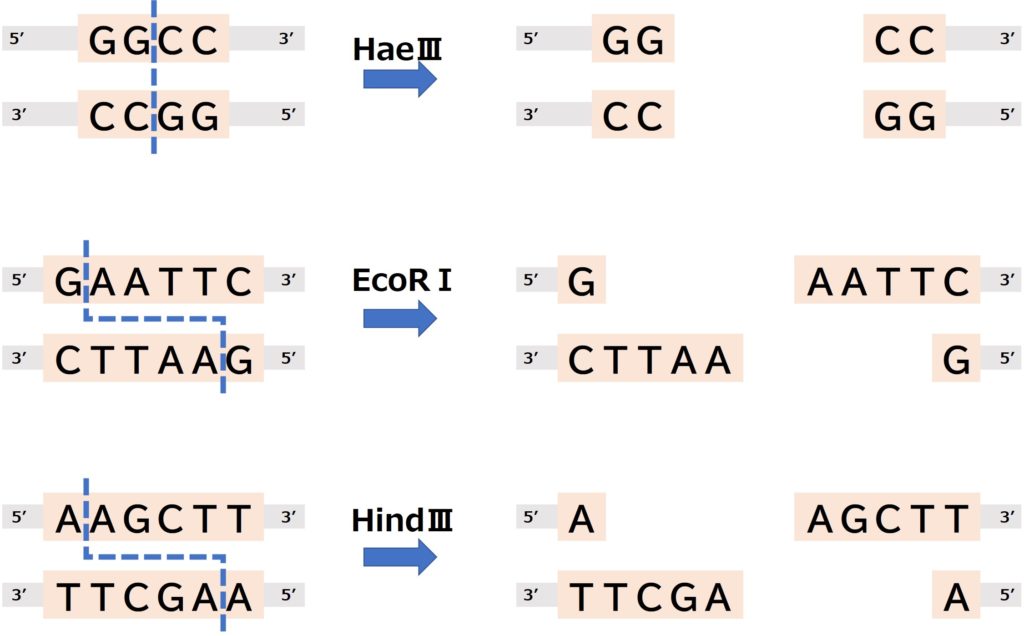
図で示したように、標的になる塩基配列は、塩基配列が真ん中を中心に点対象になっている回文構造(パリンドローム)になっていることが多いです。
例えばEcoRⅠが切断する塩基配列を見てみましょう。一方の鎖は5’末端から見るとGAATTCとなっていますが、もう一方の鎖も5’末端から見るとGAATTCと同じ配列になっています。
これが回文構造(パリンドローム)です。
たまに標的とする配列が回文構造(パリンドローム)ではない制限酵素もあります。
また、制限酵素が塩基配列をどのように切り取るかというのにもパターン2つあります。
例えばHaeⅢなどの酵素はDNAの2本鎖をまっすぐに切断し、平滑末端と呼ばれる切り口ができます。
それに対し、EcoRⅠやHindⅢという制限酵素はDNAの2本鎖を互い違いに切断し、それぞれ短い一本鎖DNAが飛び出した粘着末端と呼ばれる切り口ができます。
突出している配列が相補的な配列になっている粘着末端同士は、相補的な塩基同士が水素結合により再度つながりやすくなっています。例えば同じ制限酵素で切断してできた粘着末端は、突出している配列が相補的な配列になっているため再結合がしやすいです。
一方、平滑末端は相補的な結合ができないため再結合がしにくいという特徴があります。
実はすべての制限酵素が上記のような特徴を持っているわけではありません。制限酵素にはⅠ型、Ⅱ型、Ⅲ型、Ⅳ型があり、上記のような制限酵素はⅡ型に分類されます。
Ⅰ型の制限酵素は反応にMg2+やS-アデノシルメチオニン、ATPが必要です。Ⅰ型の制限酵素は特定の塩基配列を認識しますが、実際に切断するのはそこから数千塩基離れた領域で、しかもランダムに切断してしまいます。
Ⅱ型の制限酵素も反応にMg2+を必要とします。特定の塩基配列を認識し、認識した配列もしくはごく近くの配列を切断します。
Ⅲ型の制限酵素は、反応にMg2+とATPを必要とします。特定の塩基配列を認識し、認識した配列から25塩基対程離れた配列を切断します。
Ⅳ型の制限酵素は、メチル化されたDNAのみを切断するという特徴があります。特定の塩基配列を認識し、認識した配列から30塩基対程離れた配列を切断します。
電気泳動
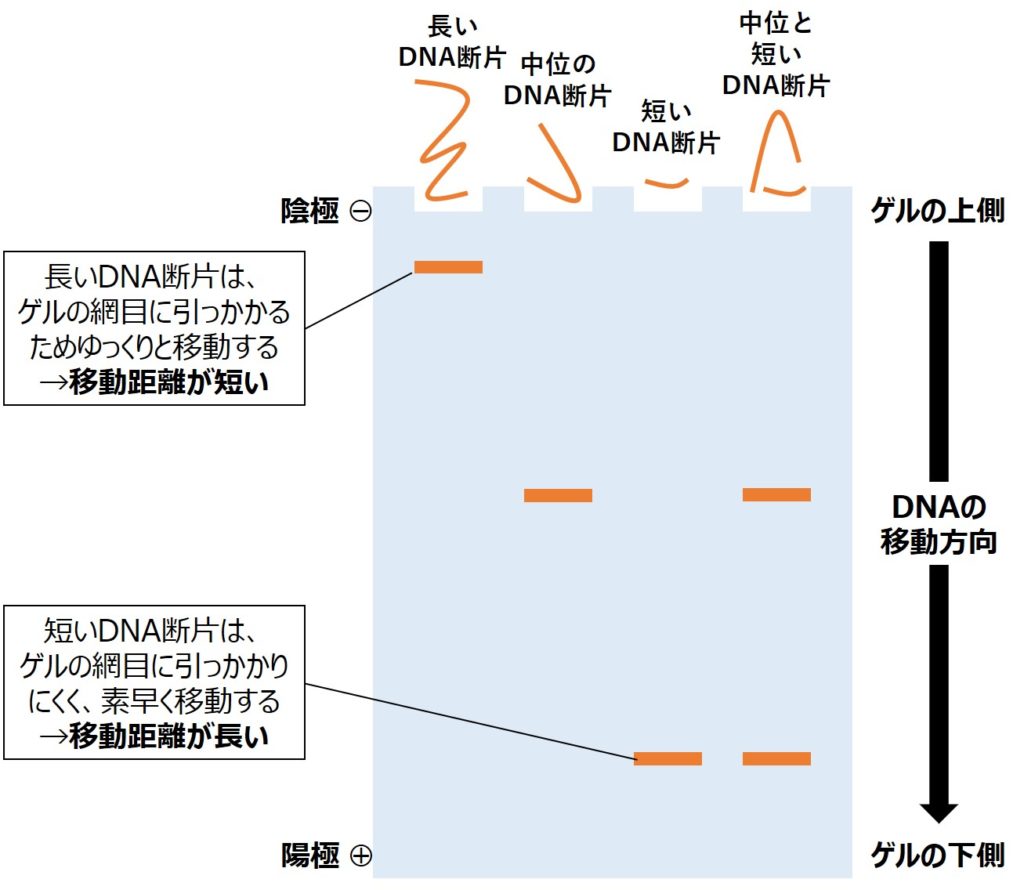
長いDNAを制限酵素で短いDNA断片に切断したら、それらのDNA断片を電気泳動という手法を用いて分けることができます。
非常に細かい網目構造をもったゲルを作製しておき、そこにDNA断片の混合物を置きます。
これに電場をかけると、DNAは負に帯電しているため、DNA断片は陽極に向かって移動していきます。
この時、長い断片はゲルの網目に引っかかりながら移動するのでゆっくりと移動します。逆に、短い断片はゲルの網目にあまり引っかからないため、素早く移動していきます。
この移動スピードの違いにより、様々な長さのDNA断片を大きさに従って分離することができ、ハシゴ上にDNA断片が並んでいきます(並んだDNA断片をバンドと呼びます)。
これを利用すると、異なる長さのDNA断片の混合物も分けることができます。図の一番右側では、中位の長さのDNA断片と短いDNA断片の混合物を電気泳動していますが、結果としてはそれぞれが別れてバンドができます。
ゲル中に分離できたDNA断片のバンドは、そのままではもちろん見ることはできません。これらのバンドを見るには、DNAを放射性同位体で標識するか、染色する必要があります。
染色する場合、DNAに結合すると紫外線によって蛍光を発する色素がよく利用されます。この色素液にゲルをさらしておき、ゲル中のDNA断片に色素を結合させます。
その後、ゲルに紫外線を当てると、DNA断片のバンドが蛍光を発するため、バンドの一が分かるという訳です。
具体例としてはエチジウムブロマイド(EtBr)があります。この物質はそのまま紫外線を当てても蛍光を発しませんが、DNA断片と結合した状態で紫外線を当てると蛍光を発するという特徴があります。ちなみにエチジウムブロマイドは、DNA断片との結合性が高いという理由から、発がん性があるとされているため、取り扱いにはちょっと気を使う試薬でもあります。
実験の例
では、どのようにこの制限酵素と電気泳動を使って実験を行っていくのか、1つの例を挙げてみましょう。
例えば、見た目に区別はつかない微生物①~③があるとしましょう。
これらはA,B,もしくはCという種類の微生物で、それぞれ遺伝子A,BもしくはCを持っています。
また、遺伝子A,BおよびCがどこでどの制限酵素で切れるかはわかっています。
遺伝子AはEcoRⅠで切断すると5000bp、3000bp、2000bpの断片に分かれ、HindⅢで切断すると6000bp、4000bpの断片に分かれます。
遺伝子BはEcoRⅠで切断すると7000bp、2000bp、1000bpの断片に分かれ、HindⅢで切断すると8500bp、1500bpの断片に分かれます。
遺伝子CはEcoRⅠで切断すると7000bp、2000bp、1000bpの断片に分かれ、HindⅢで切断すると6000bp、4000bpの断片に分かれます。
しかし、持っている遺伝子の種類によって微生物の見た目が変わるわけではないため、どの微生物がどの種類なのかが分かりません。
この時、どの微生物がどの遺伝子を持っていて、どの種類に該当するかを調べたい、といった時に、制限酵素処理と電気泳動を活用することができます。
まず、微生物①~③からDNAを抽出します。それを制限酵素であるEcoRⅠやHindⅢで切断します。切断したDNA断片を電気泳動にかけ、どのような長さのDNA断片ができているかを確認していきます。
例えば、微生物①由来のDNAをEcoRⅠで切断した場合、電気泳動の結果から、7000bp、2000bp、1000bpの長さの断片が得られるとわかったとしましょう。
そうすると、微生物①から得られたDNAは遺伝子B由来もしくは遺伝子C由来と考えられます。
どちらかを判定するために、さらにHindⅢで切断した結果も考察してみましょう。
微生物①由来のDNAをHindⅢで切断した場合、電気泳動の結果から、6000bp、4000bpの長さの断片が得られました。
すると、この微生物①から得られたDNAは遺伝子C由来であると断定できますね。
このように、制限酵素で切断した時に得られるDNA断片の長さのパターンから、得られたDNAがどのような遺伝子やDNA由来なのかを推測することができます。
まとめ
制限酵素と電気泳動、それらを組み合わせた実験の例について紹介してみました。
制限酵素処理は、今回のようなDNAのパターンを把握する以外にも、組換えDNAを作製する際などにもよく使われます。よくあるのはプラスミド(環状DNA)に遺伝子を導入するときなんかですね。
電気泳動も、実験問題などでよく問われますので、電気泳動の結果のとらえ方はよく理解しておくとよいでしょう。
実はこのあたりは、場合によっては大学院入試でも役立つかもしれない内容です。
それでは!